Masashi Yanagisawa:Deciphering the mystery of sleep: toward the molecular substrate for sleepiness
2021年11月6至7日,北京大学IDG麦戈文脑科学研究所成立十周年庆典暨北京大学脑科学国际论坛成功举办。来自美国哈佛大学、斯坦福大学、加州大学伯克利分校、英国伦敦大学学院、德国马普研究所、日本筑波大学、北京师范大学、中科院脑智卓越创新中心等国内外高校和科研机构的知名学者,从分子、细胞、环路、系统、认知、心理、计算、神经精神疾病等多个层面和角度,热情分享了他们在脑科学前沿研究的最新进展,并与线上线下观众频繁互动,在思维的碰撞中激荡出创新的火花。现陆续推出相关学术笔记,让我们重温精彩再出发!
本期推出的学术笔记,根据日本筑波大学国际综合睡眠医学研究所(International Institute for Integrative Sleep Medicine , The University of Tsukuba)Masashi Yanagisawa教授,所作的题为"Deciphering the mystery of sleep: toward the molecular substrate for sleepiness"的报告整理而成。
Masashi Yanagisawa教授作报告并与嘉宾观众交流
整理:傅升韪
审核:李毓龙
睡眠是生物之间非常保守的本能行为之一,人的一生有三分之一的时间是在睡眠中度过的。睡眠参与学习记忆的巩固等生理功能;睡眠的异常(如失眠症、发作性嗜睡病等)则会影响人们的生理及心理健康。睡眠的质量和时长受到诸多因素的影响,如外界因素包括入睡时间、睡眠环境等。那么内在的遗传因素是否可以决定睡眠的状况?如果可以,又是以怎样的方式进行调控的?对遗传因素的深入研究,将能够帮助我们更好地了解睡眠的调控机制。Masashi Yanagisawa教授分享了他的研究组基于遗传和神经环路层面研究睡眠—觉醒调控的机制成果。
1、Vglut2+神经元中Sik3突变影响睡眠表型
睡眠是否会受到特定基因的调控呢?在前期的工作中,通过对小鼠进行大规模的正向遗传筛选和基于EEG/EMG的睡眠时相监测,Yanagisawa研究组从超过8000只随机突变的小鼠中筛选出了具有异常睡眠表型的Sleepy突变个体(Funato et al., Nature, 2016)。与正常小鼠相比,Sleepy突变的小鼠每日睡眠时间显著增加,同时睡眠的需求(通过分析EEG脑电记录中与delta波相关的特征来衡量)也增加。通过定位发现嗜睡小鼠Sik3基因(Salt-Inducible Kinase 3)的第13外显子异常丢失。另一方面,在小鼠Vglut2+谷氨酸能神经元中特异性敲除Sik3的第3外显子使其功能丧失,可以观察到睡眠需求的减少以及非快速眼动睡眠(NREM)时长的减少。
为了进一步探究SIK3在哪些神经元和脑区当中产生了关键的影响,Yanagisawa研究组还构建了Sik3 exon13 flox的转基因小鼠,结合遗传学手段和利用腺相关病毒在特定脑区条件性敲除Sik3的第13外显子,将Sik3基因突变的作用区域缩小到了前侧下丘脑(anterior hypothalamus)。除了验证在Vglut2+谷氨酸能神经元中特异性敲除Sik3第13外显子能够引起NREM睡眠时长和睡眠需求的增加之外,Yanagisawa研究组还发现如果在前脑特异分布的Vglut1+谷氨酸能神经元或下丘脑特异的Foxd1+神经元中以同样的方式引入Sik3突变,分别仅会表现出睡眠时长和睡眠需求的变化,说明这一突变在上述不同脑区中可能存在不同的调控作用。
2、SIK3激酶通过调节多种突触相关蛋白磷酸化水平影响睡眠表型
Sik3作为一种蛋白激酶又是如何影响睡眠长短和睡眠需求的呢?在此后与北京生命科学研究所刘清华研究组合作的工作中,双方通过对Sleepy突变的嗜睡小鼠、野生型小鼠以及睡眠剥夺小鼠的蛋白质磷酸化组学分析共同鉴定了一系列与睡眠需求相关的磷酸化蛋白(Sleep-Need-Index PhosphoProteins, SNIPPs)(Wang et al., Nature, 2018)。相比于正常情况下,Sleepy突变个体、睡眠剥夺处理后的个体中以及药理学手段抑制SIK激酶活性的情况下,SNIPPs的磷酸化水平都会出现显著的增加。SNIPPs中有较大比例属于突触相关的蛋白,提示了SIK3激酶通过磷酸化SNIPPs调节突触功能和睡眠稳态。
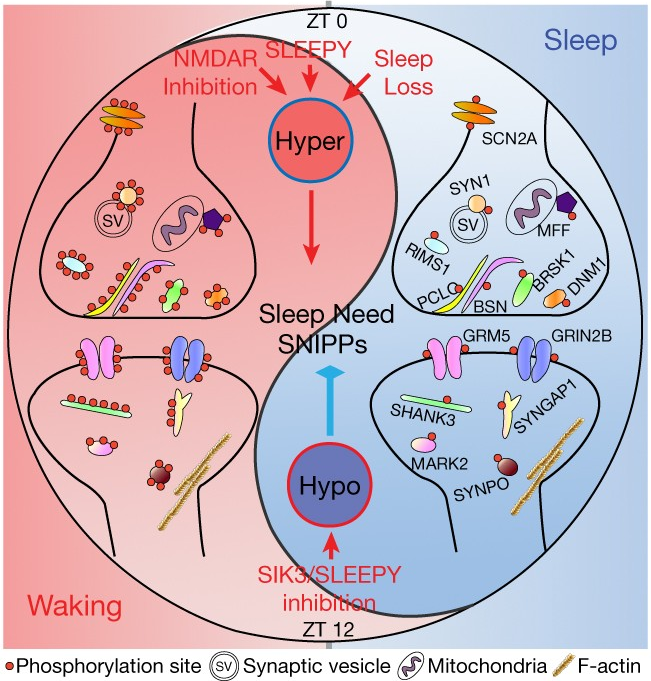
(Wang Z, et al., Nature, 2018)
3、SIK3相关胞内信号通路调节睡眠表型
作为AMPK家族蛋白激酶的成员,SIK3在分子水平上是如何调控下游通路与被调控的?Yanagisawa研究组先前发表的一项工作已经发现,在Sik3基因中缺失的第13外显子区段上的PKA磷酸化位点S551的缺失或突变都能够导致与Sleepy突变类似的表型,即NREM睡眠时长和睡眠需求的增加(Honda, et al., PNAS, 2018)。类似的,在Yanagisawa研究组先前的工作中同样观察到另一个磷酸化位点T221的磷酸化水平在睡眠剥夺情况下的上升。T221位于SIK3的激酶结构域,其磷酸化与激酶活性的增强紧密相关,在Sleepy突变个体的SIK3中引入T221A突变将激酶活性去除,能使其睡眠表型恢复到接近正常个体的水平。此外,已有报道表明LKB1(Liver Kinase B1)是SIK3上游的激酶,可能对T221的磷酸化存在调控。Yanagisawa研究组发现,在神经元中特异性敲除Lkb1后,小鼠表现出了睡眠时长和睡眠需求的减少,而这一变化在携带SIK3持续具备激酶活性的突变型个体T221E中则不存在,表明LKB1通过调节SIK3的激酶活性影响睡眠相关的表型。
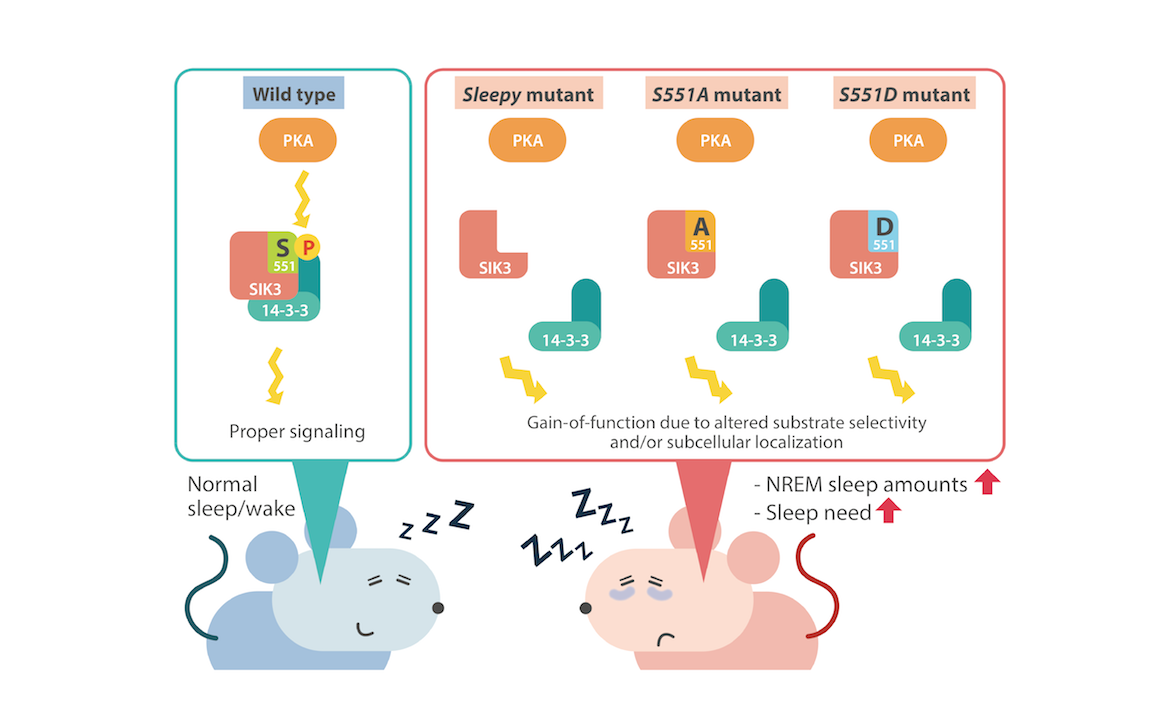
(International Institute for Integrative Sleep Medicine, University of Tsukuba)
通过之前提到的大规模遗传筛选,Yanagisawa研究组还发现了另一种具有和Sleepy表型类似的突变体,命名为Sleepy2,其对应的突变存在于Hdac4基因(Histone Deacetylase 4)中,而HDAC4已经被报道存在于SIK3激酶的下游。那么SIK3是否通过调控下游的HDAC4从而调控睡眠呢?Yanagisawa研究组首先验证了SIK3上的Sleepy突变会导致HDAC4上S245位点的过度磷酸化,从而减少HDAC4对下游转录因子MEF2的抑制。随后,他们发现S245A突变的小鼠表现出了符合预期的睡眠表型的恢复,表明SIK3能够通过作用于HDAC4的特定磷酸化位点实现对睡眠稳态的调控。至此,通过大规模遗传筛选和生理生化实验的验证,Yanagisawa研究组这一部分尚未发表的工作表明LKB1-SIK3-HDAC4通路参与睡眠稳态的调控。
4、便携式EEG的开发和推广
除了上述与睡眠相关的基础研究外,Yanagisawa教授还关注人类睡眠相关的临床健康问题。他提到了目前睡眠障碍患者的就医困难和繁琐诊断过程,诊断中存在患者自主报告的不准确性,以及由此带来的诸多诊疗中的实际障碍等。为了更切实地解决这类问题,Yanagisawa教授和合作者们已经初步开发出适用于人类患者的更加便携的EEG记录设备。新的设备有望实现更加普及和方便的睡眠质量测量和诊断,从而惠及更多目前尚待接受诊疗的睡眠障碍患者。
参考文献
Funato, H., Miyoshi, C., Fujiyama, T., Kanda, T., Sato, M., Wang, Z., ... & Yanagisawa, M. (2016). Forward-genetics analysis of sleep in randomly mutagenized mice. Nature, 539(7629), 378-383.
Wang, Z., Ma, J., Miyoshi, C., Li, Y., Sato, M., Ogawa, Y., ... & Liu, Q. (2018). Quantitative phosphoproteomic analysis of the molecular substrates of sleep need. Nature, 558(7710), 435-439.
Honda, T., Fujiyama, T., Miyoshi, C., Ikkyu, A., Hotta-Hirashima, N., Kanno, S., ... & Yanagisawa, M. (2018). A single phosphorylation site of SIK3 regulates daily sleep amounts and sleep need in mice. Proceedings of the National Academy of Sciences, 115(41), 10458-10463.
