饶毅:指示和控制睡眠需求的化学修饰
2021年11月6至7日,北京大学IDG麦戈文脑科学研究所成立十周年庆典暨北京大学脑科学国际论坛成功举办。来自美国哈佛大学、斯坦福大学、加州大学伯克利分校、英国伦敦大学学院、德国马普研究所、日本筑波大学、北京师范大学、中科院脑智卓越创新中心等国内外高校和科研机构的知名学者,从分子、细胞、环路、系统、认知、心理、计算、神经精神疾病等多个层面和角度,热情分享了他们在脑科学前沿研究的最新进展,并与线上线下观众频繁互动,在思维的碰撞中激荡出创新的火花。
本期推出的学术笔记,根据北京大学IDG麦戈文脑科学研究所长饶毅教授,所作的题为“Sleep Need: Indicator and Controller”的学术报告整理而成。
至此,论坛所有报告的相关学术笔记全部推送完毕。感谢关注,薪火相传,创新致远,未来可期!
撰稿:夏冰心、黄燕云
审核:李扬
饶毅实验室近年的研究主线之一是睡眠的分子机制。自2017年以来,他们发表了5篇睡眠分子机理的论文,其中4篇通过果蝇遗传学和分子生物学(参考文献1到4)、1篇老鼠分子生物学(参考文献5)。
饶毅教授报告了实验室尚未发表的研究成果,用生物化学、分子生物学和遗传学揭示睡眠需求的分子机理。他们发现,一个蛋白质分子的一个位点的磷酸化修饰能够同时指示和控制睡眠需求。
饶毅教授首先简要介绍了睡眠机理的相关研究。
1982年,Alexander Borbély提出睡眠有两种调控:C过程和S过程。C过程就是昼夜节律,已经有很多分子机理的研究,而对S过程的理解很有限。
S过程是睡眠的自稳态(homeostasis):睡眠多导致睡眠需求减少,睡眠少导致睡眠需求增加。
至今,没有分子或细胞满足既能够指示、又能够控制睡眠需求的标准。
日本科学家Masashi Yanagisawa实验室在2016发文指出(参考文献6),盐诱导激酶3(Salt Inducible Kinase 3,SIK3)的突变可影响睡眠。当 SIK3 的S551位点突变或包含这个位点的片段被删除后,SIK3的活性会增加,导致小鼠产生sleepy的表型,睡得更多。S551本身被蛋白激酶A(PKA)所磷酸化。
Yanagisawa实验室在其它文献中也发现了SIK1和SIK2在相应的PKA位点有相似突变情况下,小鼠睡眠也会增多,睡眠需求增加。
但是在SIK1、2、3的研究中,它们的突变都是功能增加的突变(gain of function,GOF),没有功能缺失的突变(loss of function,LOF)。用GOF获得的结果表明分子功能提高后的影响,这些影响可以是生理性、也可以不是生理性,GOF不能证明分子对于生理的必要性。也就是说,SIK1、2、3迄今的GOF突变,提示它们可以影响睡眠,但不证明它们参与控制正常的生理睡眠。
另外,SIK3的S551位点磷酸化水平,不随睡眠需求而改变,这样S551的磷酸化不指示睡眠需求。

图1 Masashi Yanagisawa实验室发文指出SIK3的功能增加突变可以影响睡眠
现在,饶毅实验室在小鼠体内把SIK1敲除,图2左显示敲除后小鼠脑中SIK1 mRNA水平下降,图2右展示了EEG的NREM delta density,这是普遍公认的小鼠睡眠需求的测量标准。SIK1敲除后小鼠的睡眠需求与野生型相比无显著差异。可以看到,缺乏SIK1,不改变24小时的睡眠需求。所以,虽然Yanagisawa实验室显示SIK1功能GOF突变改变了睡眠需求,而饶毅实验室发现SIK1的LOF突变不改变睡眠或睡眠需求,证明SIK1并不参与调控生理的睡眠需求。
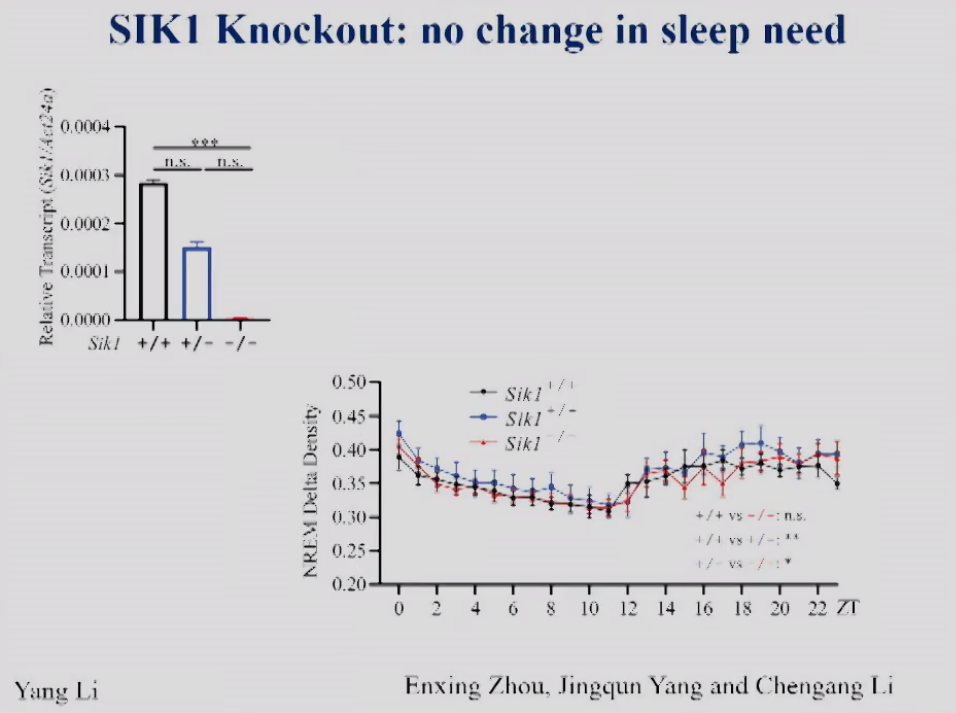
图2 SIK1敲除小鼠脑组织中SIK1 mRNA的相对表达量和NREM delta density。
饶毅实验室也敲除了SIK2,也就是制造其LOF突变。图3显示,缺乏SIK2后,老鼠的睡眠需求也无没有改变。这些结果说明,虽然Yanagisawa实验室显示SIK2功能GOF突变改变了睡眠需求,而饶毅实验室发现SIK2的LOF突变不改变睡眠或睡眠需求,证明SIK2并不参与调控生理的睡眠需求。
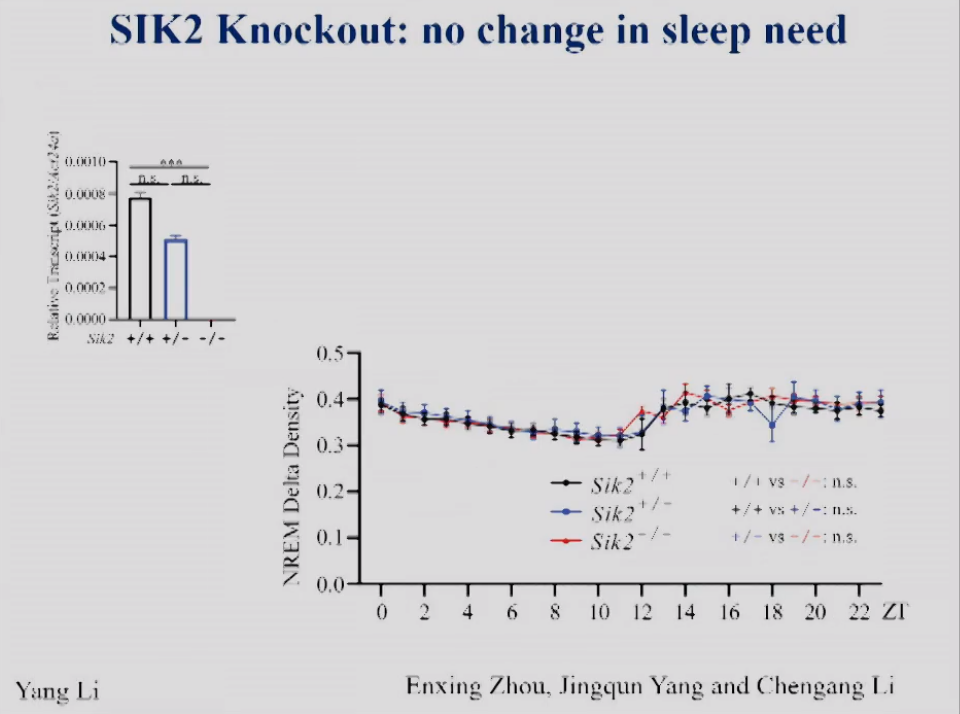
图3 SIK2敲除小鼠脑组织中SIK2 mRNA的相对表达量和NREM delta density。
为了研究SIK3 LOF在睡眠调控中的作用,饶毅实验室先制作了SIK3敲除小鼠,但是大部分敲除小鼠都是胚胎致死,只有很少量的小鼠进行后期的睡眠分析。为此,他们一面分析SIK3敲除的小鼠,一面又构建了SIK3敲低小鼠。
图4左显示,在SIK3敲低小鼠脑中SIK3 mRNA的水平降低。图4右显示,在SIK3 敲低小鼠中,NREM delta density显著低于野生型,证明了SIK3是睡眠需求生理必须的。
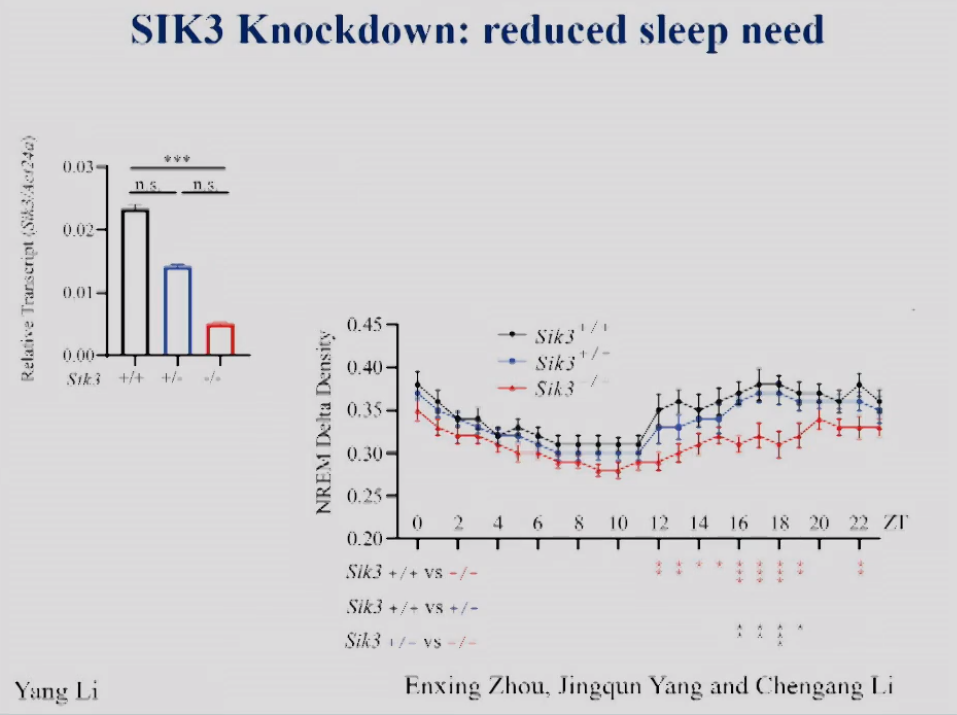
图4 SIK3敲低小鼠脑组织中SIK3 mRNA的相对表达量和NREM delta density。
图5表格显示了不同时间点小鼠的睡眠需求。从表格中可以看出,在野生型小鼠中,睡眠需求随着时间而变化,ZT16、ZT17的睡眠需求和ZT9的睡眠需求是有显著差异的。而在敲低小鼠中睡眠需求变化有了显著的延迟,直到ZT20才有了一点增加。
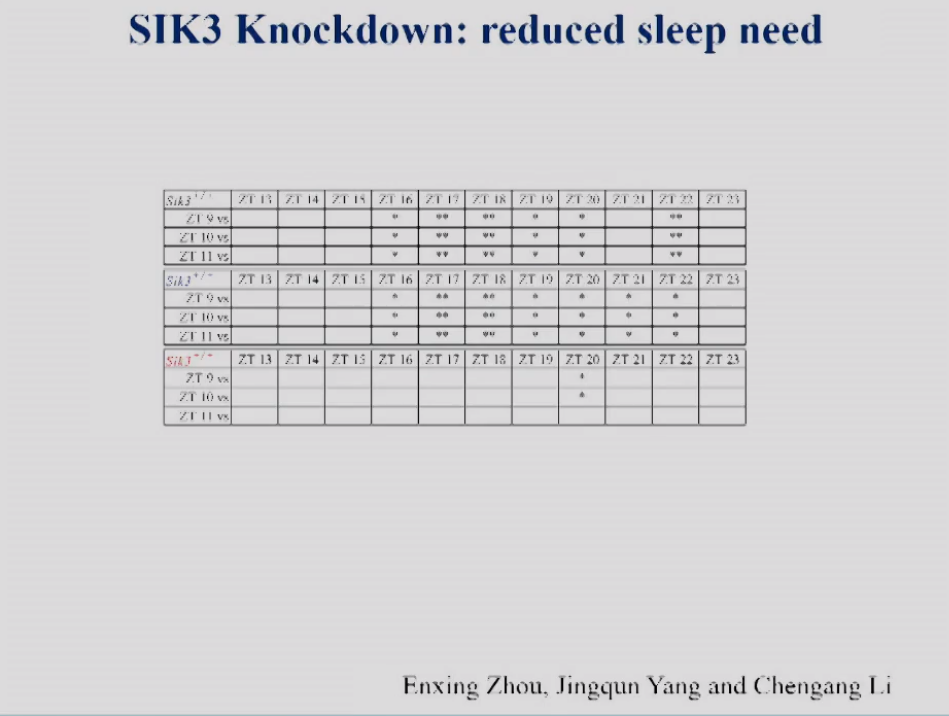
图5 不同时间点的小鼠的NREM delta power density的统计比较。
另一个证明控制睡眠需求的标准是睡眠剥夺实验。
正常情况下,小鼠被剥夺睡眠之后,有反弹--睡眠需求增加、睡眠增多。与对照组小鼠相比,SIK3敲低小鼠无论是杂合突变还是纯合突变,其在睡眠剥夺之后的睡眠反弹都显著降低(图6)。
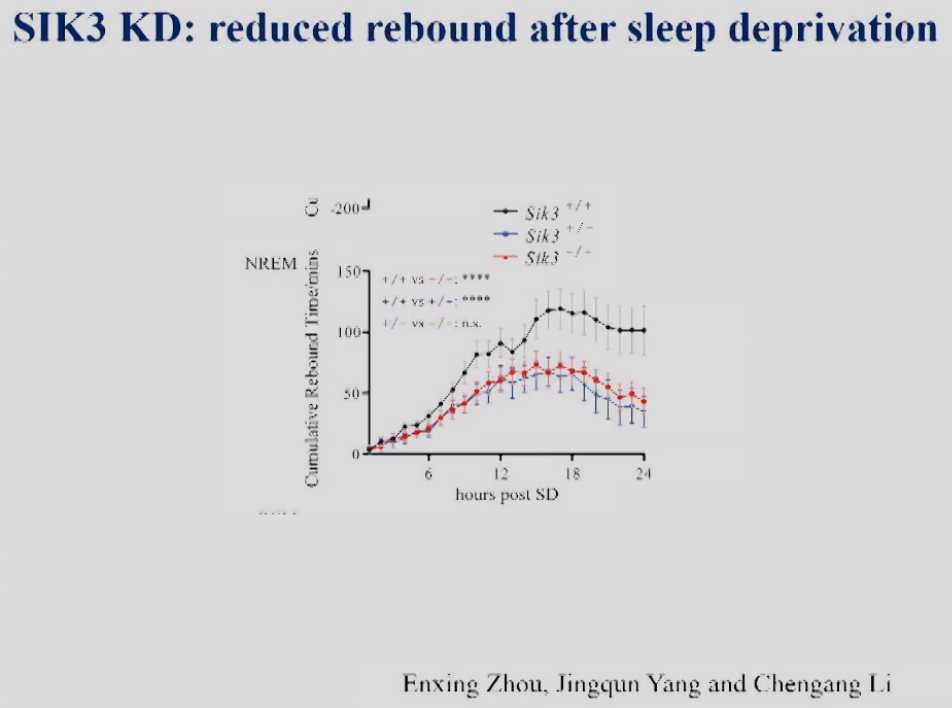
图6 SIK3敲低小鼠在SD后6小时的累计NREM睡眠增加。
以上结果证明SIK1、2不是睡眠需求所必需的,而SIK3为生理学睡眠需求所必需。
Yanagisawa实验室发现SIK3 551位点磷酸化,但其磷酸化程度不会随着睡眠需求而变化。此外,他们通过质谱发现另一个T221位点睡眠剥夺之后,磷酸化程度增加。
饶毅实验室研究了SIK3的磷酸化与其激酶的催化活性。图7左磷酸酶处理去除了SIK3的磷酸化后,SIK3对下游蛋白HDAC4的磷酸化作用减弱,说明SIK3的激酶活性下降。图7右显示T221位点突变为A后,不再被其上游激酶所磷酸化,SIK3对其下游靶点HDAC4的磷酸化作用也显著降低了,说明T221位点的磷酸化对于SIK3的激酶活性是必需的。

图7 左:SIK3的催化活性,右:SIK3 WT和T221A突变体蛋白的酶活性。
饶毅实验室还发现T221位点的磷酸化对于维持SIK3蛋白的稳定性也十分重要。图8上显示HEK细胞转染SIK3后,在0h用蛋白合成抑制剂CHX处理细胞,可见2小时后蛋白有部分降解,而接下来的22小时基本保持稳定。而细胞转染T221A突变的SIK3后,24小时内蛋白很快降解(图8下显示)。这证明了SIK3 T221磷酸化对维持SIK3蛋白稳定性是必要的。饶毅实验室还发现,当221位点的T突变A后,鼠脑中SIK3蛋白水平也下降了(图8右)。所以,在细胞和鼠脑中,SIK3的T221对SIK3蛋白稳定都是必要的。
从两组生物化学实验得出结论,SIK3的T221对SIK3的催化功能和SIK3蛋白稳定性都是必要的。

图8 左:SIK3蛋白在HEK293T细胞中的降解分析,右:SIK3 点突变小鼠大脑中SIK3 T221磷酸化水平和SIK3蛋白表达。
饶毅实验室用特异识别SIK3 T221磷酸化的抗体检测其在体内磷酸化状况。图9左在细胞中验证了抗体的特异性,当正常转染SIK3时,该抗体可以识别到特异条带,而加入磷酸酶处理之后条带信号减弱,说明识别到的是磷酸化条带;而当SIK3 T221位点突变为A之后,该磷酸化抗体不能在对应位置检测到特异条带,说明该抗体是特异识别磷酸化的SIK3 T221位点。他们在小鼠中同样检测了该抗体的特异性。图9中显示,SIK3敲除之后,SIK3蛋白缺失,该抗体也未检测到信号条带;图9右显示在SIK3 T221A的突变小鼠脑中,虽然SIK3的蛋白降低,但是该磷酸化抗体依然未有检测到信号条带。这些结果表明这一抗体在体内和体外都很特异。
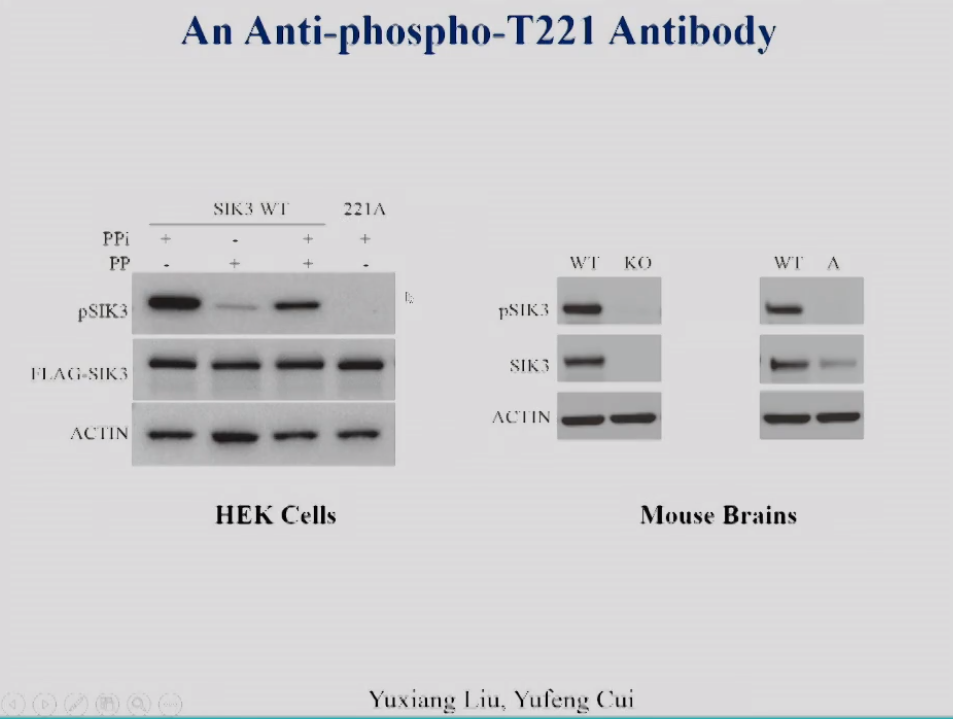
图9 左:抗SIK3 pT221单抗在HEK293T细胞中的特异性,右:SIK3敲除和点突变小鼠脑匀浆中的SIK3 pT221和SIK3蛋白表达。
饶毅实验室在连续36个小时里,每3个小时采集12个小鼠大脑,总共13个时间点。跑蛋白凝胶后,用抗磷酸化T221抗体检测。
他们发现,在睡眠开始时,SIK3和SIK3 T221的蛋白水平非常高,睡眠过程中SIK3蛋白水平和SIK3 T221磷酸化水平都下降。老鼠醒后的12小时,SIK3 T221磷酸化蛋白和SIK3总蛋白都会增加,再过12小时也会看到相同的周期变化。反之,而对生物钟重要的蛋白PER2,每个时间点都不同于SIK3,周期也不同,每个时间点PER2 662位点磷酸化水平也不同,因此这可以显示SIK3磷酸化水平与睡眠需求相关。
睡眠需求越多,SIK3蛋白和SIK3 T221磷酸化就越多。睡眠需求越少, SIK3蛋白和SIK3 T221磷酸化就越少。
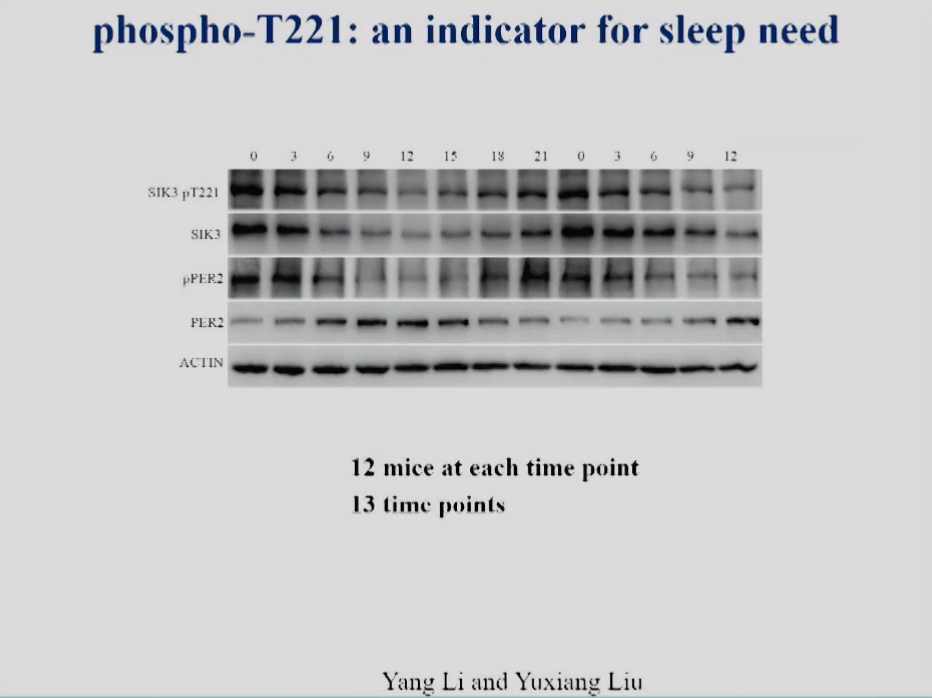
图10 小鼠大脑SIK3蛋白水平,T221磷酸化水平,PER2蛋白水平和PER2 S662磷酸化水平的时间谱(各时间点n=12)。
饶毅实验室还重复了Masashi Yanagisawa在睡眠剥夺方面的工作。图11显示,当小鼠睡眠6小时后,SIK3水平下降,T221磷酸化水平下降。但在同样的6小时内,如果人为剥夺不让老鼠睡觉,那么T221磷酸化水平和SIK3总蛋白水平就不降低。相比之下,不仅肌动蛋白actin,还有ERK/JNK等蛋白激酶的磷酸化水平都没有改变。所以,T221磷酸化无论是在睡眠剥夺的情况下,还是在自然的睡眠—觉醒周期中,都能指示睡眠需求。
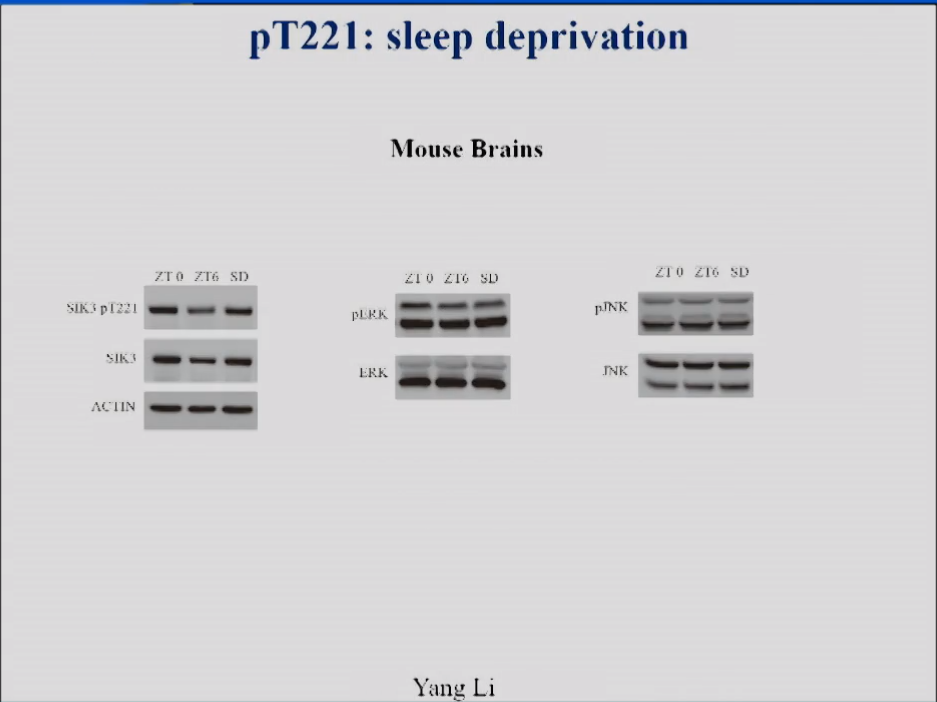
图11 睡眠剥夺对小鼠大脑SIK3和T221磷酸化的影响。
为了研究T221位点的睡眠调控作用,饶毅实验室制备了SIK3的T221A突变小鼠。图12显示,作为睡眠需求主要的指标-NERM Delta density,T221A纯合突变体显著低于野生型。
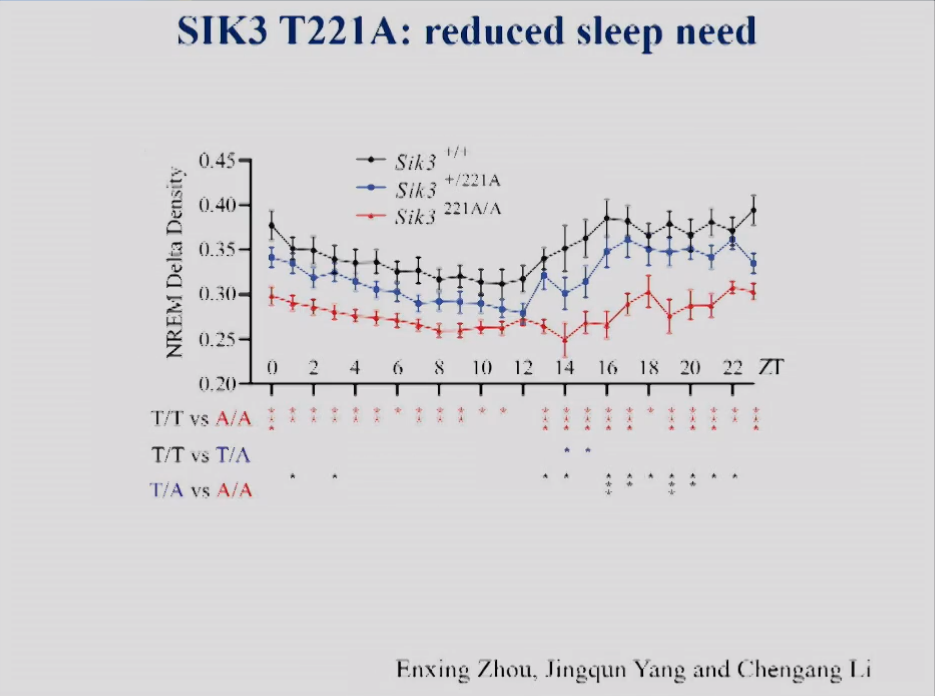
图12 T221A纯合突变体和野生型小鼠的NERM Delta density。
图13显示,在小鼠睡眠剥夺实验中,野生型小鼠睡眠剥夺后睡眠会反弹。在杂合子小鼠和纯合T221A突变小鼠中,睡眠剥夺后的反弹显著低于野生型小鼠。
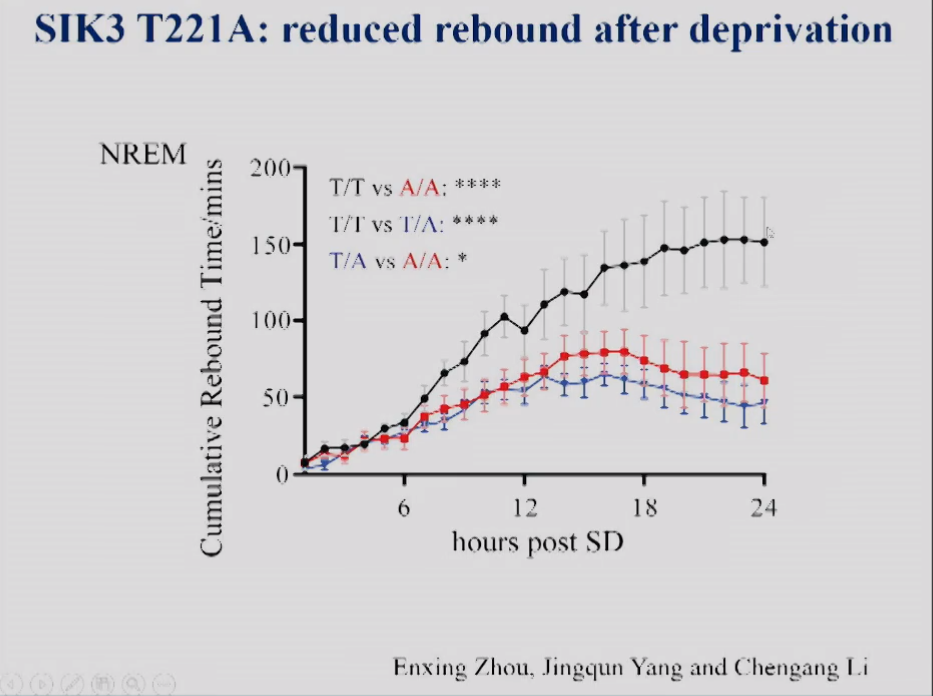
图13 SIK3 T221A小鼠在SD后6小时累积NREM睡眠增加。
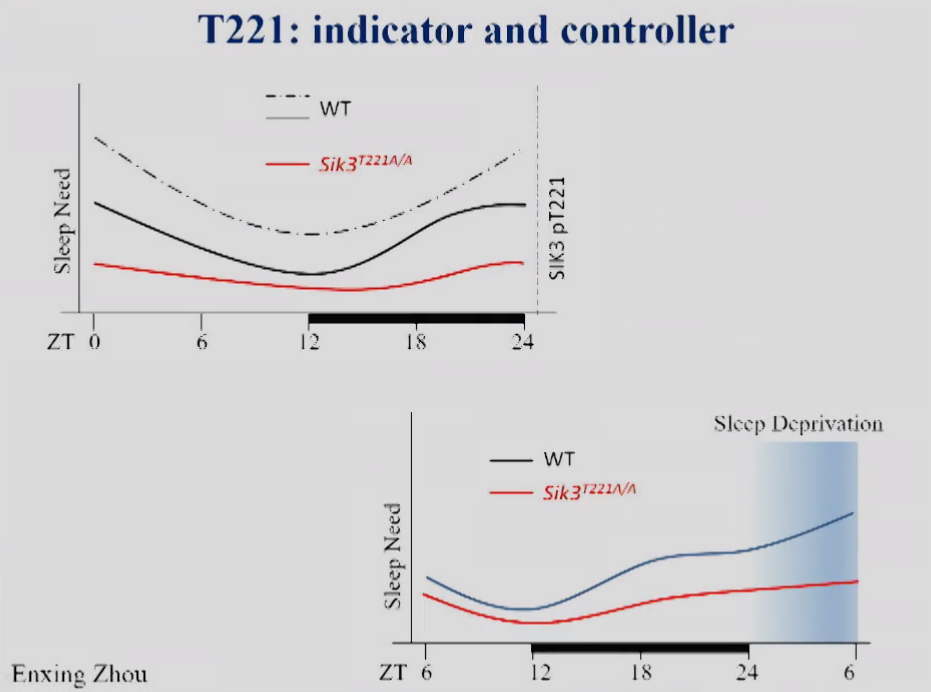
图14 SIK3-T221磷酸化如何指示和控制小鼠睡眠需求的示意图。
饶毅实验室发现SIK3蛋白中T221位点的磷酸化既指针、又控制睡眠需求。他们提出了图14的模型:SIK3的T221在过程S的作用。
在此之前,只能通过测量脑电图中的NERM delta density来测量小鼠的睡眠需求。而现在可以据此发现,通过检测单个分子的单点磷酸化来测量睡眠需求。

饶毅教授与Masashi Yanagisawa教授在线交流
在其后的问答互动环节,饶毅教授与Masashi Yanagisawa教授,就LKB1与SIK3二者之间的关系,在线进一步展开探讨。饶毅教授指出,LKB1于2003年被几个小组发现,可以磷酸化AMPK,而SIK3 属于AMPK相关激酶。2004年《EMBO杂志》的论文显示LKB1 可以磷酸化所有 14 种与 AMPK 相关的激酶。饶毅实验室在果蝇和小鼠中对LKB1进行了敲除,以表明LKB1影响睡眠。现在复杂的是,在进行生化实验的过程中发现,来自HEK 细胞免疫沉淀的LKB1可以磷酸化SIK3,细菌表达的重组LKB1可以直接磷酸化重组AMPK,但是细菌表达的重组LKB1不能磷酸化重组SIK3。所以饶毅实验室花了几年时间,发现除了LKB1之外还有另外二十几种蛋白激酶,细菌重组表达的时候,也可以磷酸化SIK3和AMPK。所以,AMPK和SIK3上游的情况比想象的要复杂很多。遗传学和分子生物学对睡眠研究的重要性不言而喻,而生物化学和化学生物学对睡眠研究,很可能非常重要。
参考文献
1. Qian YJ, Cao Y, Deng BW, Yang G, Li J, Xu R, Zhang D, Huang J and Rao Y (2017). Sleep homeostasis regulated by 5HT2b receptor in a small subset of neurons in the dorsal fan-shaped body of Drosophila. eLife 6:e26519.
2. Deng BW, Li Q, Liu XX, Cao Y, Li BF, Qian YJ, Xu R, Mao RB, Zhou EX, Zhang WX, Huang J and Rao Y (2019) Chemoconnectomics: mapping chemical transmission in Drosophila. Neuron 101:876-893.
3. Dai XHM, Zhou EX, Yang W, WX Zhang and Rao Y (2019). D-Serine promotes sleep through the NMDA receptor in Drosophila melanogaster. Nature Communications 10:1986.
4. Dai XHM, Zhou EX, Yang W, Deng BW, Li Q, Liu XX, Zhang WX and Rao Y (2021). Molecular resolution of a behavioral paradox: sleep and arousal are regulated by distinct acetylcholine receptors in different neurons of Drosophila. Sleep 10, 1093.
5. Zhang X, Yan HM, Huang ZL and Rao Y (2018). Independence of 5HT involvement in sleep and arousal from thermoregulation in mice. Molecular Pharmacology 93:657-664.
6. Funato H., Miyoshi C., Fujiyama T., Kanda T., Sato M., Wang Z., et al. (2016). Forward-genetics analysis of sleep in randomly mutagenized mice. Nature 539 378–383.
