Alice Ting: Optogenetic and chemogenetic technologies for probing molecular and cellular interactions
2021年11月6至7日,北京大学IDG麦戈文脑科学研究所成立十周年庆典暨北京大学脑科学国际论坛成功举办。来自美国哈佛大学、斯坦福大学、加州大学伯克利分校、英国伦敦大学学院、德国马普研究所、日本筑波大学、北京师范大学、中科院脑智卓越创新中心等国内外高校和科研机构的知名学者,从分子、细胞、环路、系统、认知、心理、计算、神经精神疾病等多个层面和角度,热情分享了他们在脑科学前沿研究的最新进展,并与线上线下观众频繁互动,在思维的碰撞中激荡出创新的火花。现陆续推出相关学术笔记,让我们重温精彩再出发!
本期推出的学术笔记,根据斯坦福大学(Stanford University)Alice Ting教授,所作的题为“Optogenetic and chemogenetic technologies for probing molecular and cellular interactions”的报告整理而成。她在报告中分享了三种新兴探测分子间细胞间相互作用的光遗传和化学遗传技术。

Alice Ting教授作报告并与嘉宾观众交流
撰稿:程思慧
审核:苗成林
1
FLiCRE(Fast Light and Calcium-regulated Expression),一种光控钙整合器,可以快速标记、记录并控制在研究任务中激活的细胞亚群,通过结合流式筛选及单细胞测序技术,还可进一步揭示这些细胞亚群的转录组特征。
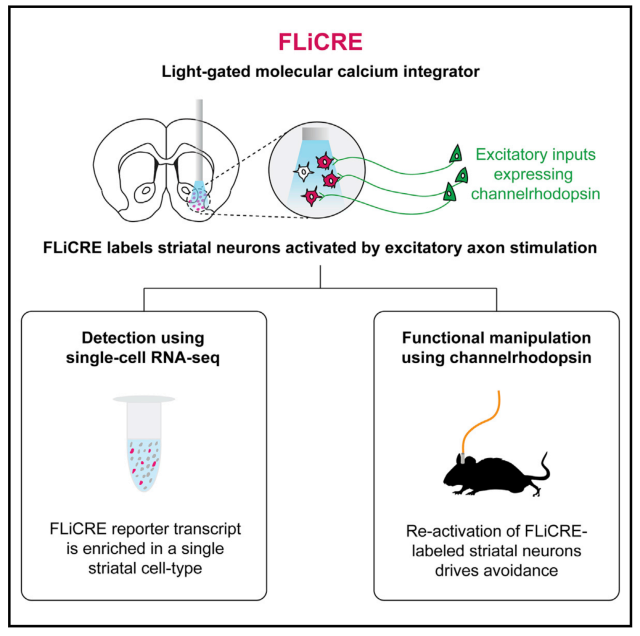
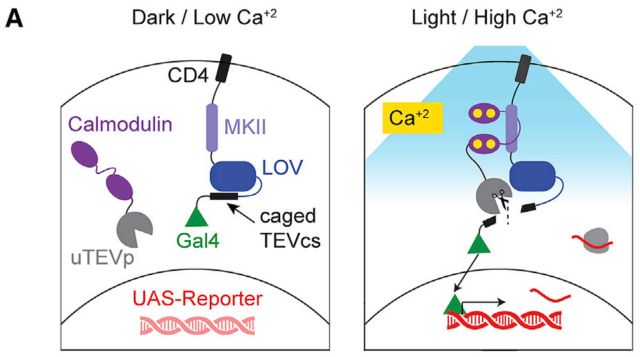
FLiCRE由两种主要的分子组分组成,一是在胞浆中钙调蛋白 (CaM) 和蛋白酶uTEVp的融合蛋白,另一个是带有一系列分子 (CD4、MKII、LOV和caged TEVcs) 锚定在细胞膜上的转录因子Gal4。只有当细胞被473 nm激光激发且处于高钙状态时,CaM和MKII结合,uTEVp剪切caged TEVcs,才能释放转录因子Gal4进核,驱动下游报告基因(比如TRE-mCherry)的表达。现有的标记技术,比如c-fos-tTA mice、TRAP2 mice等,都需要几小时才能被激活,而FLiCRE仅需要60s,快速又敏感。
Alice Ting团队首先在HEK293T 细胞中开发并验证了此工具的可行性,又在体外培养的皮层神经元中鉴定了FLiCRE工具的高敏感性和可逆性,最后在小鼠体内进行了应用。他们将FLiCRE注射到小鼠的伏隔核(nucleus accumbens, NAc),用473 nm激光激发NAc的同时,用光遗传手段激活NAc的上游脑区——前额叶皮层(prefrontal cortex, PFC)的神经元,从而NAc中接受PFC投射的细胞亚群被报告基因mCherry所标记。为了进一步揭示这类细胞亚群的特征,他们使用单细胞测序的方法,发现NAc中表达mCherry的是一类高表达Tac1和Calb1的特定细胞亚群。那么这类细胞亚群是否在小鼠的回避行为中发挥功能呢?Alice Ting团队将FLiCRE工具中的报告基因替换成带有bReaChES的兴奋性视蛋白,从而可以用橙光激活NAc中接受PFC神经元投射的细胞亚群。在实时条件性位置偏好实验中,他们发现激活NAc中的这类细胞亚群时,小鼠表现出回避行为,说明可以利用FLiCRE控制NAc中驱动回避行为的特殊细胞亚群[1]。
Alice Ting团队还在不断改进FLiCRE工具,开发出了scFLARE (A single-chain calcium integrator),将FLiCRE的两种组份简化成一种结构,不仅背景噪音更低,并且使用时只需要注射一种AAV即可[2],具有广泛的应用价值。
2
HiLITR (High-throughput Localization Indicator with Transcriptional Readout),将蛋白共定位转化成膜锚定转录因子的水解释放,从而启动报告基因的表达,可以用于高通量筛选影响膜定位蛋白的调控因子。
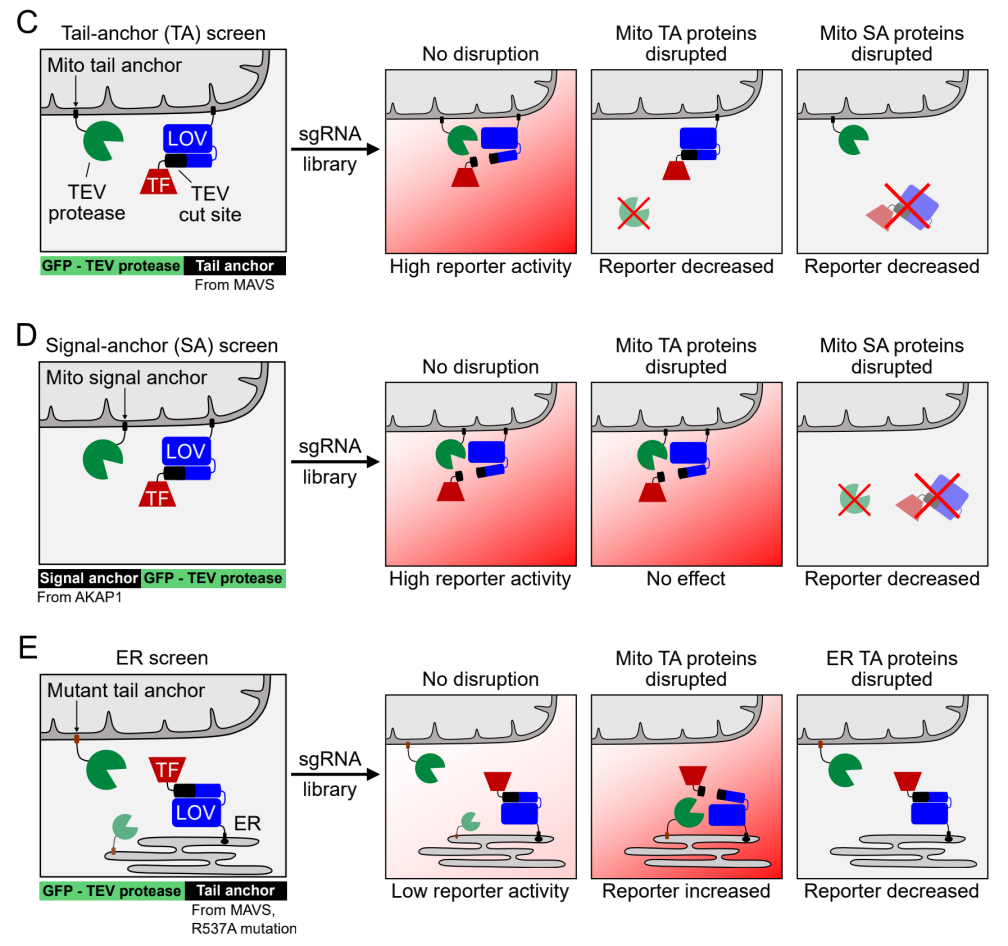
特定的蛋白在正确的时间被运输到正确的位置对于生物系统中的信号转导和调节都是必不可少的,而高通量基因筛选可以为探索蛋白运输的分子机制提供很大的帮助。然而只有当蛋白错误定位能够产生简单而稳健的的细胞选择表型时,目的基因才能被有效地筛选出来。HiLITR是一种用于高通量筛选影响膜定位尾锚定(tail-anchored, TA)蛋白基因的分子生物学技术。Alice Ting团队将TA膜定位蛋白和蛋白酶(TEV protease)相连,同时将目标膜的信号锚定(signal-anchored, SA)膜定位蛋白,连接LOV,TEVcs和转录因子。只有在473 nm激光激发下,并且两个蛋白在膜上共定位,TEV蛋白酶才能发挥作用剪切TEVcs,释放转录因子,从而启动荧光蛋白的表达。

在高通量筛选中,若某基因影响此TA蛋白的膜定位,则荧光相较于对照组会产生变化,进而可以通过流式细胞术将这些细胞筛选出来(TA筛选)。同时为了避免筛选到影响SA而非TA(或SA和TA都影响)的假阳性基因,Alice Ting团队将SA膜定位蛋白和TEV蛋白酶相连进行筛选,从而通过荧光强度改变确定假阳性基因(SA筛选)。另外,由于损坏或者非靶向线粒体的TA蛋白可能重新回到内质网膜,他们还设计了内质网筛选(ER筛选),将内质网、高尔基体膜的标记蛋白连接LOV,TEVcs和转录因子,将一个突变的线粒体TA蛋白(mTA*)和TEV蛋白酶融合,该突变蛋白增加了内质网、高尔基膜错误定位的倾向。ER筛选不仅可以排除影响内质网及高尔基体膜定位蛋白的假阳性基因,还有助于筛选调节内质网膜蛋白的基因。随后他们利用HiLITR和高通量筛选,鉴定了影响线粒体和内质网TA蛋白运输的基因。结果发现,SUMOE1中SAE1的缺失会导致许多线粒体TA蛋白的错误定位,此外还证明了EMC10在内质网膜复合物中的独特调控作用[3]。HiLITR通过将基因的影响转化为直观的荧光信号指标,降低了高通量筛选的复杂程度,提高筛选效率,扩大了通过遗传扰动筛选技术来研究的生物过程范围。
3
TPL(Tandem proximity labeling),邻近标记,将目的蛋白与具有邻近标记功能的酶进行融合,在活细胞中,该酶催化可扩散反应的发生,将邻近的蛋白标记上生物素,之后通过质谱或测序可以鉴定出与目的蛋白相互作用的分子。
TPL利用了诸多工具酶,例如过氧化物酶(APEX)、辣根过氧化物酶(HRP)或生物素连接酶(TurboID、miniTurbo)等,这些酶通过基因编辑的手段连接到研究者感兴趣的蛋白质上。在生物体内,这些蛋白连接的酶将周围的惰性小分子底物转化为具有高度活性且寿命极短的反应物,例如APEX催化产生自由基或TurboID催化产生酯类化合物。这些催化产生的反应物从酶活性位点扩散到与之相邻近的其他分子上,对邻近分子进行共价标记。标记范围的半径由反应物的半衰期和环境中猝灭剂浓度所决定,在活细胞中,实验确定的标记半径一般在1-10 nm内。实验中用到的底物分子通常包含生物素,以便可以使用链霉亲和素磁珠富集被标记的邻近分子,并通过质谱或核酸测序对其进行鉴定[4]。
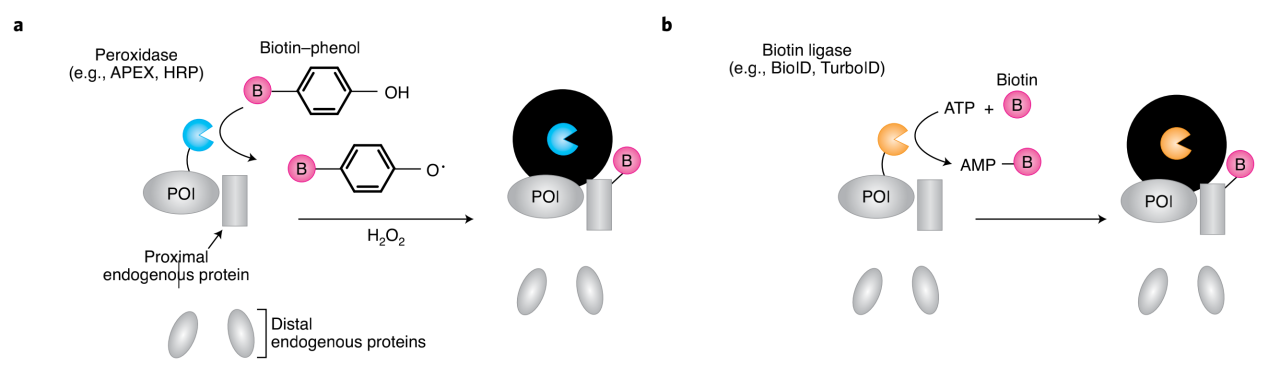
通过不同TPL工具酶的定位和表达,此方法可用于从整个细胞,到细胞器,再到大分子复合物等不同尺度的邻近标记检测。Alice Ting实验室研究出TurboID和miniTurbo两种新型工具酶,极大提升催化活性,室温条件下仅需10min即可标记成功,同时在果蝇和线虫活体中也能有效工作[5]。
参考文献
[1] Kim, C. K., Sanchez, M. I., Hoerbelt, P., Fenno, L. E., & Ting, A. Y.. (2020). A molecular calcium integrator reveals a striatal cell type driving aversion. Cell, 183(7).
[2] Sanchez, M. I., Nguyen, Q. A., Wang, W., Soltesz, I., & Ting, A. Y.. (2020). Transcriptional readout of neuronal activity via an engineered Ca2+-activiated protease. Proceedings of the National Academy of Sciences, 117(52), 202006521.
[3] Coukos, R. W., Yao, D., Sanchez, M. L., Strand, E. T., & Ting, A. Y.. (2021). An engineered transcriptional reporter of protein localization identifies regulators of mitochondrial and er membrane protein trafficking in high-throughput screens. eLife Sciences.
[4] Wei, Q., Cho, K. F., Cavanagh, P. E., & Ting, A. Y.. Deciphering molecular interactions by proximity labeling. Nature Methods.
[5] Li, J., Han, S., Li, H., Udeshi, N. D., Svinkina, T., & Mani, D. R., et al. (2020). Cell-surface proteomic profiling in the fly brain uncovers wiring regulators. Cell, 180(2).